library( tidyverse )
set.seed( 1325 )
tibble( seed_weight = rnorm( n=50, mean=50, sd=10 ) ) %>%
mutate( plant_height = rnorm( n=50, mean=30, sd=5 ) + 0.1*seed_weight ) -> plant_dataLineare Regression
Beispieldaten
Wir möchten wissen, ob das Gewicht eiens Samens einen Einfluss auf das Wachstum einer Pflanze hat. Wir wiegen 50 Samen, pflanzen sie ein, warten 4 Wochen, und messen dann die Höhe der Pflanze.
Der folgende Code erstellt Beispiel-Daten
Unsere Samen wiegen also im Schnitt 50 mg mit Standardabweichung 10 mg. Die Pflanzen werden im Schnitt 30 cm hoch mit einer (vom Gewichtd es Samens unabhägigen) Standardabweichung von 5 cm. Dazu kommt ein Effekt von Effekt von 0.03 cm extra für jedes mg Samen-Gewicht.
Hier ist ein Plot:
ggplot(plant_data) + geom_point( aes( x=seed_weight, y=plant_height ) )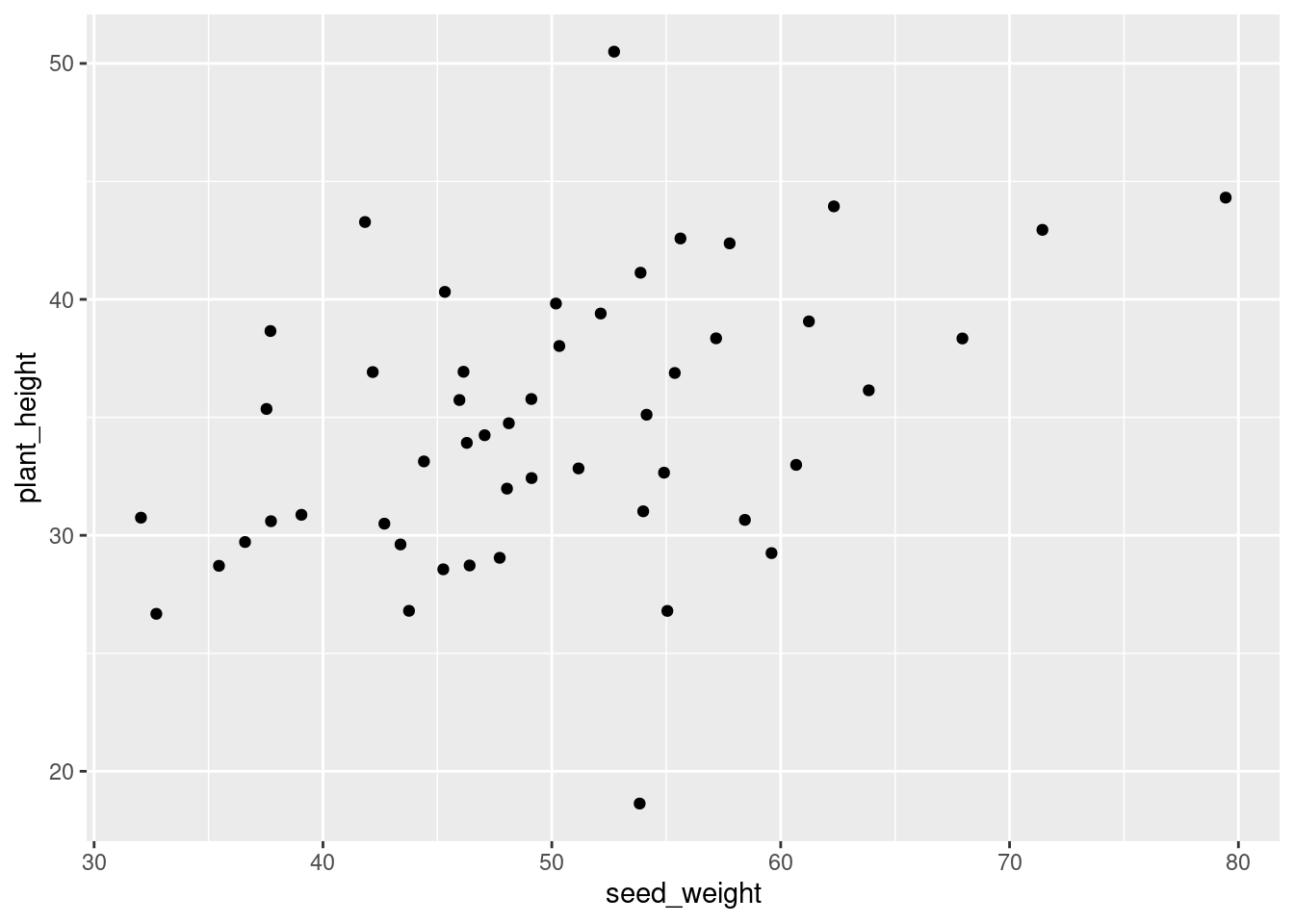
Fitten eines linearen Modells
fit <- lm( plant_height ~ seed_weight, plant_data )
fit
Call:
lm(formula = plant_height ~ seed_weight, data = plant_data)
Coefficients:
(Intercept) seed_weight
22.3204 0.2482 Die Werte der Koeffizienten können wir aus dem fit-Objekt entnehmen:
coef(fit)(Intercept) seed_weight
22.3203968 0.2481968 Unser Fit ergibt also folgende Formel für den fitted value für die Höhe \(y_i\) (in cm) von Pflanze \(i\):
\[ \hat y_i = 31.4 + 0.067 x_i, \] wobei \(x_i\) das Gewicht des Samens in mg ist.
Regressionsgerade
Wir zeichnen dies in unseren Plot ein:
ggplot(plant_data) +
geom_point( aes( x=seed_weight, y=plant_height ) ) +
geom_abline( intercept = coef(fit)[["(Intercept)"]], slope = coef(fit)[["seed_weight"]] )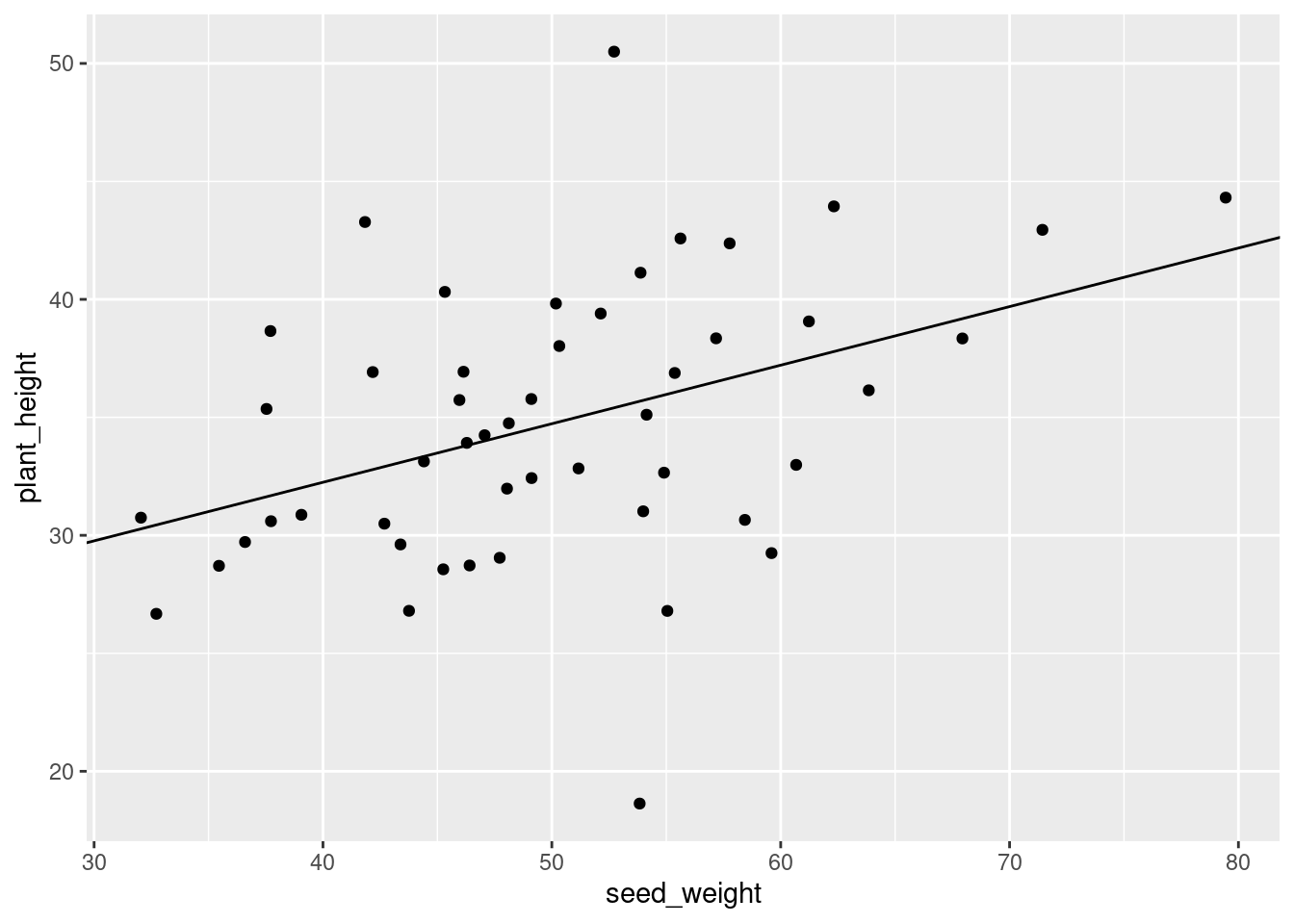
Diese Linie (“Ausgleichsgerade”, “Regressionsgerade”) ist den Daten angepasst (fitted to the data), in dem Sinne, dass die Residuen-Quadratsumme \(\text{RSS} = (y_i - \hat y_i)^2\) minimiert wurde.
Signifikanz
Wieviel Varianz wird erklärt, und ist das signifikant?
summary(fit)
Call:
lm(formula = plant_height ~ seed_weight, data = plant_data)
Residuals:
Min 1Q Median 3Q Max
-17.0506 -3.0742 -0.0505 3.1982 15.0927
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 22.32040 4.02404 5.547 1.22e-06 ***
seed_weight 0.24820 0.07886 3.147 0.00283 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 5.408 on 48 degrees of freedom
Multiple R-squared: 0.1711, Adjusted R-squared: 0.1538
F-statistic: 9.905 on 1 and 48 DF, p-value: 0.002831Ja.
Die Steigung ist aber deutlich zu groß. Wie immer sollten wir uns aber nicht auf die Punktschätzung verlassen, sondern ein Konfidenzintervall berechnen. Die Tabelle oben enthält bereits eine Standardfehler für den Koeffizienten, und die Funtion confint berechnet ein 95%-Konfidenzintervall:
confint( fit ) 2.5 % 97.5 %
(Intercept) 14.22952988 30.4112637
seed_weight 0.08963603 0.4067577Korrelationskoeffizient
Das Bestimmtheitsmaß \(R^2\), dass wir oben erhalten haben, ist das Quadrat des Pearson-Korrelationskoeffizienten:
cor( plant_data$seed_weight, plant_data$plant_height )[1] 0.4135942cor( plant_data$seed_weight, plant_data$plant_height )^2[1] 0.1710602Zur Erinnerung, die Formel für den Pearson-Korrelationskoeffizient einer Liste von Werte-Paaren \((x_i,y_i)\) mit Mittelwerten \(\mu_x\) unf \(\mu_y\):
\[ R = \frac{\sum_i (x_i-\mu_x)(y_i-\mu_y)}{\sqrt{\sum_i (x_i-\mu_x)^2\sum_i(y_i-\mu_y)^2}}\]
Code zum Testen
Hier ist der Code nochmal zusammen gestellt. Durch mehrfaches ausführen können wir sehen, wie das Ergebnis variieren kann.
tibble( seed_weight = rnorm( n=50, mean=50, sd=10 ) ) %>%
mutate( plant_height = rnorm( n=50, mean=30, sd=5 ) + 0.1*seed_weight ) -> plant_data
fit <- lm( plant_height ~ seed_weight, plant_data )
ggplot(plant_data) +
geom_point( aes( x=seed_weight, y=plant_height ) ) +
geom_abline( intercept = coef(fit)[["(Intercept)"]], slope = coef(fit)[["seed_weight"]] )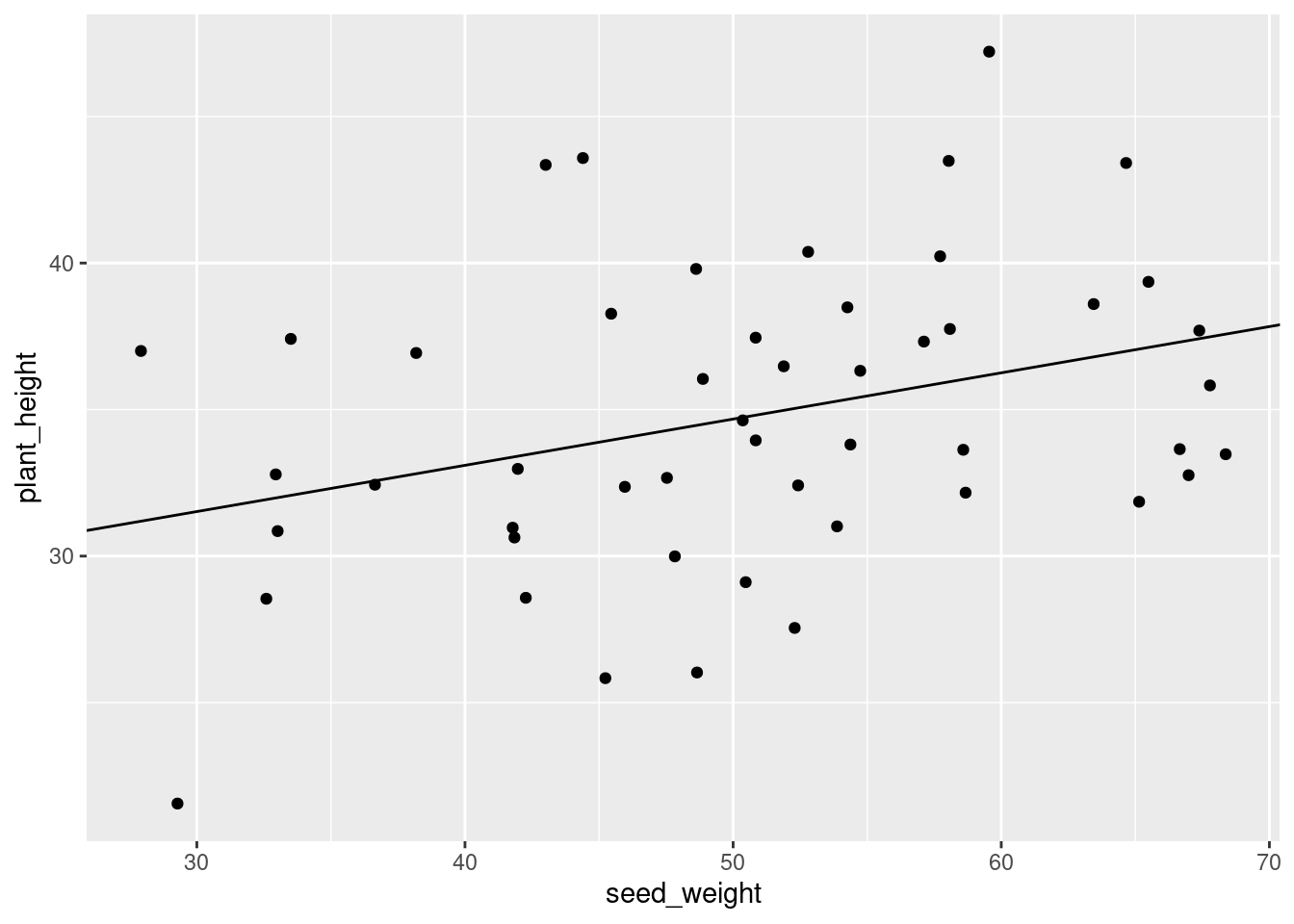
coef(fit)(Intercept) seed_weight
26.7861615 0.1577994 confint(fit) 2.5 % 97.5 %
(Intercept) 20.0805513 33.491772
seed_weight 0.0281757 0.287423summary(fit)
Call:
lm(formula = plant_height ~ seed_weight, data = plant_data)
Residuals:
Min 1Q Median 3Q Max
-9.8517 -3.5876 -0.2836 3.0153 11.0303
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 26.78616 3.33507 8.032 1.97e-10 ***
seed_weight 0.15780 0.06447 2.448 0.0181 *
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 4.889 on 48 degrees of freedom
Multiple R-squared: 0.111, Adjusted R-squared: 0.09244
F-statistic: 5.991 on 1 and 48 DF, p-value: 0.01809Wachstum von Kindern
Wir laden die NHANES-Daten und filtern auf die Kinder (bis 15 Jahre):
read_csv( "data_on_git/nhanes.csv" ) %>%
filter( age < 16, !is.na(height) ) -> nhanes_childrenNun fitten wir ein lineares Modell:
fit <- lm( height ~ age, nhanes_children )
fit
Call:
lm(formula = height ~ age, data = nhanes_children)
Coefficients:
(Intercept) age
81.799 6.058 Wir plotten dies:
ggplot( nhanes_children ) +
geom_point( aes( x=age, y=height, col=gender ), size=.3, position=position_jitter(width=.2,height=0) ) +
geom_abline( intercept = coef(fit)["(Intercept)"], slope=coef(fit)["age"] )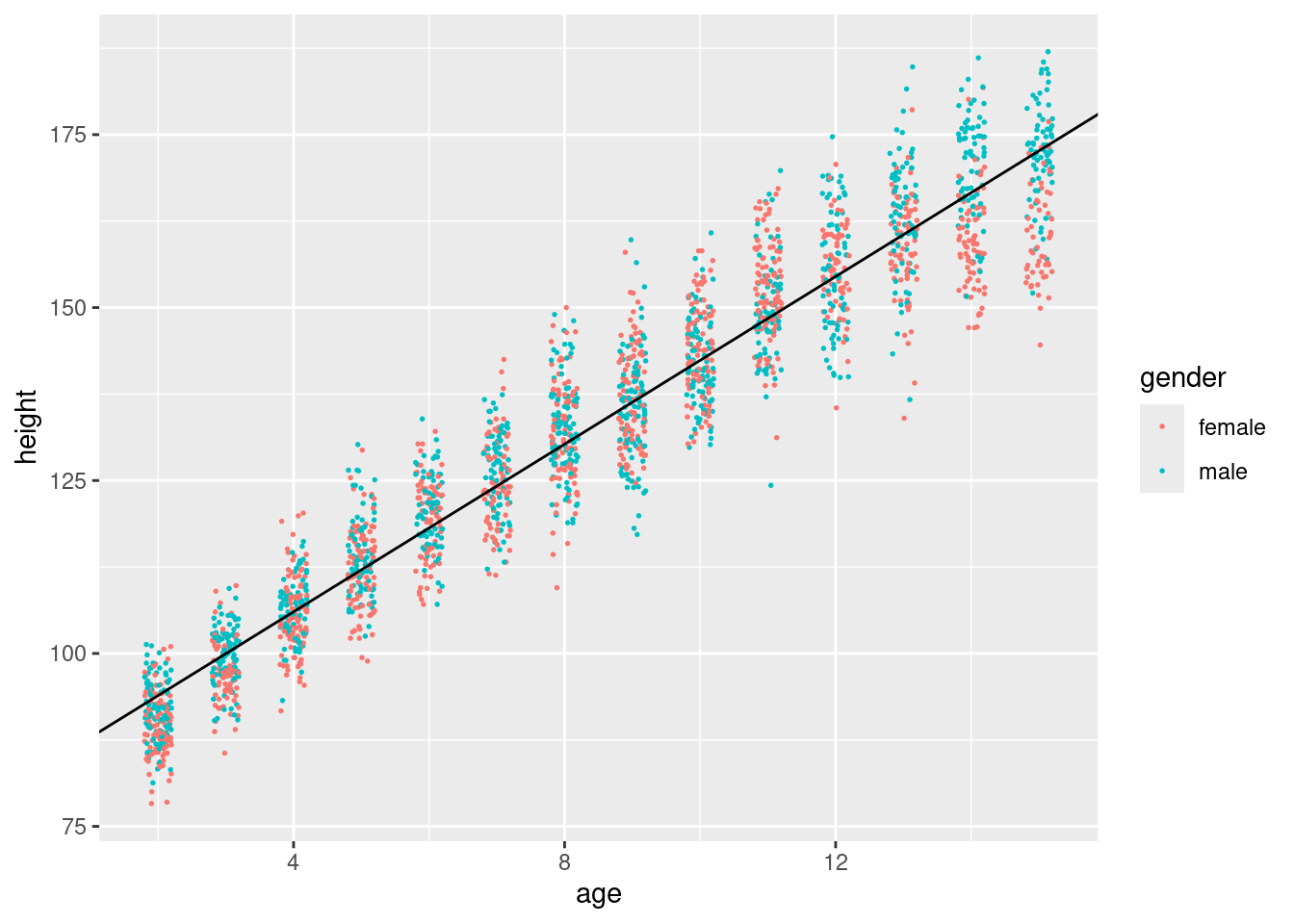
Kategorische und kontinuierliche Variable
Zurück zum Beispiel der Samen. Nun wurde die Hqlfte der Samen vorher getrocknet. Dies verringert den Wuchs:
set.seed( 1327 )
tibble( seed_weight = rnorm( n=50, mean=50, sd=15 ) ) %>%
mutate( dry = rep( c( "no", "yes" ), each=25 ) ) %>%
mutate( plant_height = rnorm( n=50, mean=30, sd=5 ) +
0.3*seed_weight + ifelse( dry=="yes", -.5, 0 ) ) -> plant_dataWir möchten wissen, ob die Trocknung einen Unterschied macht.
Erster Versuch: t-Test
t.test( plant_height ~ dry, plant_data )
Welch Two Sample t-test
data: plant_height by dry
t = 2.0686, df = 43.475, p-value = 0.04457
alternative hypothesis: true difference in means between group no and group yes is not equal to 0
95 percent confidence interval:
0.09254813 7.20059264
sample estimates:
mean in group no mean in group yes
46.76732 43.12075 Signifikant, aber nur gerade eben.
Ein Plot zeigt, warum:
ggplot(plant_data) + geom_point( aes( x=seed_weight, y=plant_height, col=dry ) )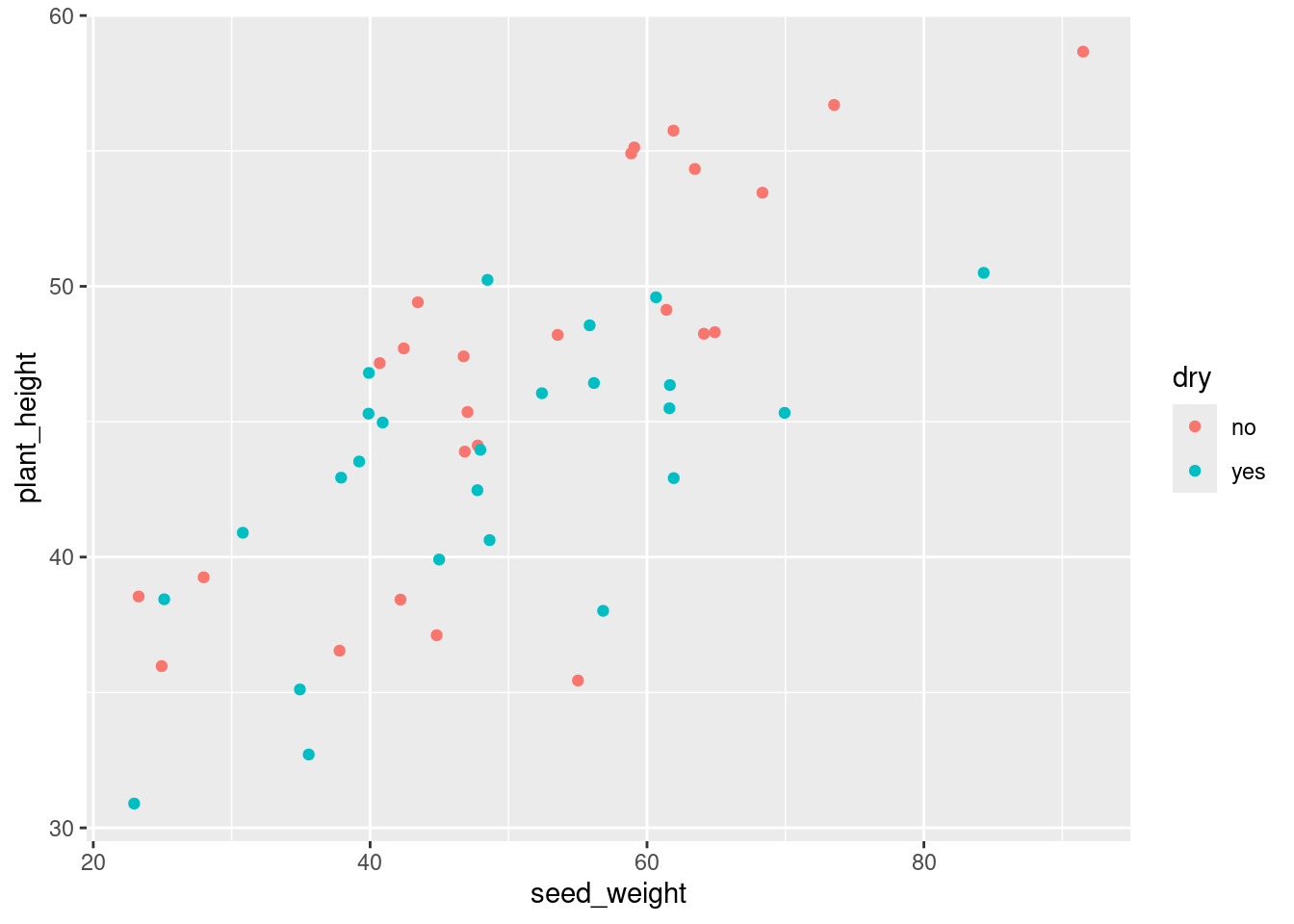
Wenn wir erst seed_weight “heraus-regressieren” (to regress out), und die Residuen vergleichen würden, sollten wir ein klareres Ergebnis bekommen:
anova( lm( plant_height ~ dry + seed_weight, plant_data ) )Analysis of Variance Table
Response: plant_height
Df Sum Sq Mean Sq F value Pr(>F)
dry 1 166.22 166.22 8.8477 0.004621 **
seed_weight 1 981.63 981.63 52.2515 3.683e-09 ***
Residuals 47 882.97 18.79
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1