suppressPackageStartupMessages( library(tidyverse) )
plate_cols <- tibble(
plate_column = 1:12,
used = c( FALSE, rep(TRUE, 10), FALSE ),
content = c( "empty", rep( "co-culture", 8 ), rep( "target cells only", 2 ), "empty" ),
subject = c( NA, rep( c( "C1", "P1", "C2", "C3" ), 2 ), rep( NA, 3 ) ),
aphidicolin = c( NA, rep( TRUE, 9), FALSE, NA ),
n0_target_cells = c( 0, rep( 12000, 10), 0 ),
n0_t_cells = c( 0, rep( 12000, 4), rep( 24000, 4), rep( 0, 3 ) ),
) Übung: Ein “Killing-Assay” zur Untersuchung von Immunschwäche
In der folgenden Hausaufgabe analysieren Sie Daten aus einem echten Experiment aus laufender Forschung. Sie dient also dazu, Ihnen einen Einblick zu bieten, wie R in der Praxis eingesetzt wird.
Die Aufgabe ist etwas länger. Schauen Sie, wie weit Sie am Wochenende kommen, und laden Sie den Teil auf Moodle hoch. Den Rest machen wir in der Übung am Dienstag gemeinsam.
Unser Beispiel
Wir nutzen hier Daten, die mir eine Kollegin von der Universität Helsinki zur Verfügung gestellt hat. Sie beschäftigt sich mit genetischen Krankheiten.
Der Patient P1 leidet an einer angeborenen Immunschwäche.
Bei einer DNA-Sequenzierung wurde eine seltene Mutation gefunden, die ein Gen funktionslos macht, das in der Aktivierung der zytotoxischen T-Zellen eine Rolle spielt.
Es wird also vermutet, dass die zytotoxischen T-Zellen dieses Patienten eine im Vergleich zu gesunden Menschen verringerte Fähigkeit haben, pathologische Zellen zu bekämpfen.
Daher sollen die zytotoxischen T-Zellen von Patient P1 in sog. Killing-Assays mit zytotoxischen T-Zellen von 3 gesunden Probanden (C1, C2 und C3) verglichen werden. Die 3 Vergleichpersonen (“control subjects”) haben dasselbe Geschlecht und ein ähnliches Alter (“gender- and age-matched controls”).
Killing-Assays
Zytotoxische T-Zellen (CD8-T-Zellen) dienen dazu, virus-infizierte Zellen zu erkennen und zu eliminieren.
Bei einem “Killing Assay” wird die Effektivität von zytotoxischen T-Zellen gemessen, in dem sie zusammen mit geeigneten “Target-Zellen” inkubiert werden. Man misst, wie viele der Targetzellen von den T-Zellen eliminiert werden – oder genauer: man zählt, wie viele der Targetzellen nach einer bestimmten Zeit noch übrig sind und nicht eliminiert wurden.
Als Target-Zellen verwendet man gerne immortalisierte Zell-Linien, die man mit anti-CD3 belegt, damit sie von den T-Zellen als Ziel gesehen werden.
Die Target-Zellen enthalten ein Transgen für GFP (green flourescent protein), damit sie grün floureszieren und so leicht unter dem Mikoskop erkannt und gezählt werden können.
Vorbereitung der Proben
Von Patient und Vergleichsprobanden wurde peripheres Blut entnommen, und es wurden mittels Ficoll-Gradient-Zentrifugierung die mononuklearen weißen Blutzellen extrahiert (PBMC = peripheral blood mononuclear cells).
Um genügend Material zu haben, wurden die T-Zellen vorab in einer Zellkultur vermehrt (Nährmedium mit anti-CD3, anti-CD28 und IL2, um die T-Zellen zu aktivieren und zur Teilung anzuregen). Nach 10 bis 13 Tagen standen 108 Zellen zur Verfügung.
Andere Zellen als die zytotoxische T-Zellen wurden mittels MACS (magnet-assisted cell sorting) entfernt, d.h., magnetische Kügelchen, die mit Antikörpern gegen Marker der anderen PBMC-Zelltypen beschichtet sind, ziehen diese Zellen heraus.
Als Target-Zellen wurde die murinen Zelllinie P815 verwendet. Die Targetzellen exprimieren GFP. Sie wurden mit anti-CD3 belegt, damit die T-Zellen sie als “schädlich” betrachten. Außerdem wurde Aphidicolin, ein Proliferations-Hemmer, zugegeben, um die Targetzellen an der Vermehrung zu hindern.
Für das eigentlich Assay wurde eine 96-Well-Platte verwendet, in der in jedes Well eine andere Kombination aus T-Zellen und Target-Zellen eingebracht wurde. Das Schema, wie die Proben auf der Platte verteilt wurden (das. sog. Platten-Layout) ist weiter unten beschrieben.
Automatisches Mikroskop im Inkubator
Zur Durchführung des Assays wurde ein Gerät verwendet, bei dem ein automatisches Mikroskop in einem Inkubator verbaut ist. Das Gerät wird mit einer Mikrotiter-Platte mit Proben bestückt und kann so programmiert werden, dass es in regelmäßigen Abständen von jedem Well der Platte mikroskopische Bilder aufnimmt.
Hier ist ein typisches Bild, wie es von dem Gerät aufgenommen wurde:
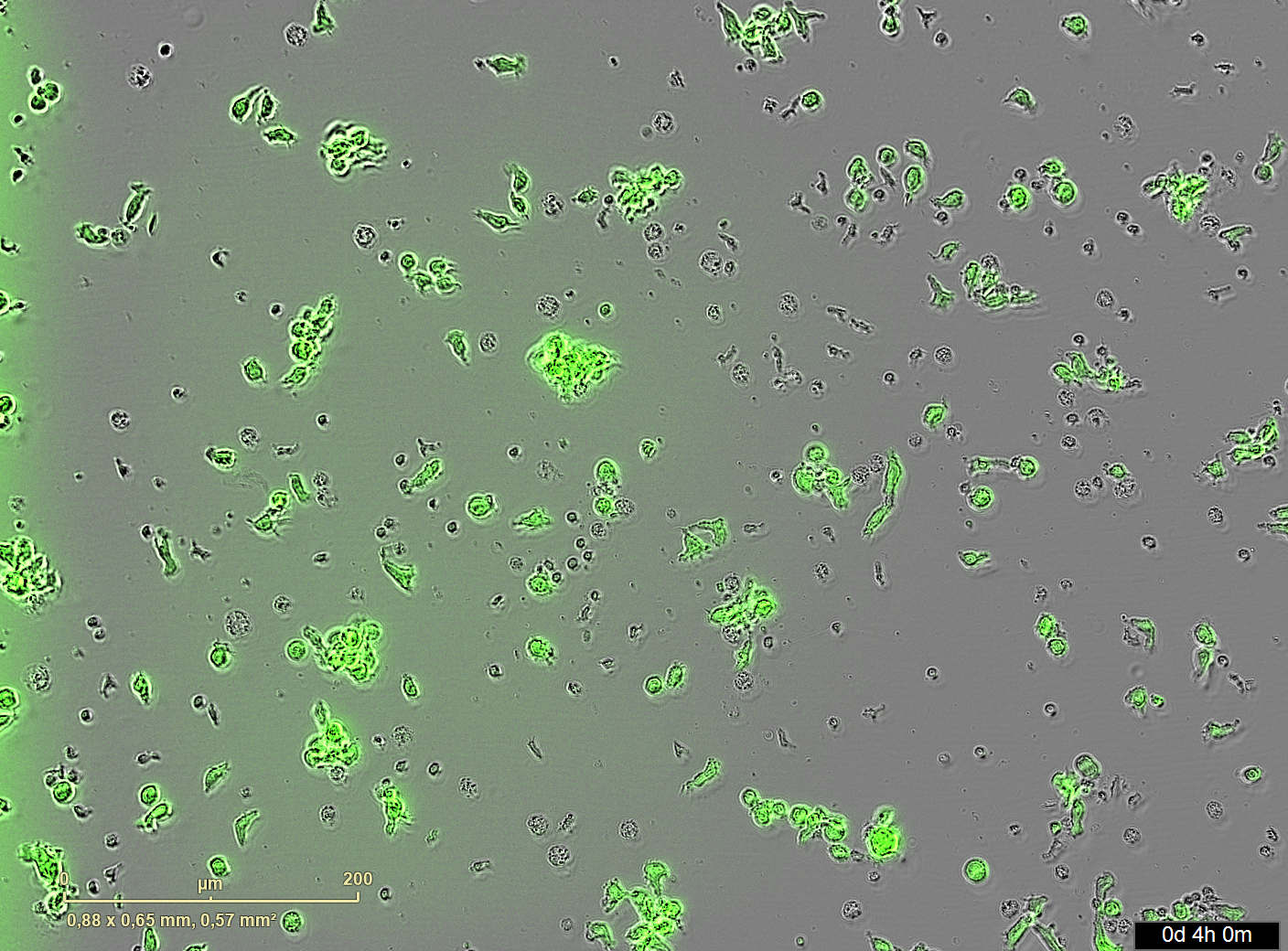
Die großen grünen Zellen sind die Target-Zellen, die kleineren nicht-grünen die T-Zellen.
Dieses Bild ist von Well F6 (siehe unten), aufgenommen nach 4 h Inkubationszeit.
Das Gerät zählt automatisch die grünen Zellen und erstellt am Schluss eine Tabelle mit diesen Zahlen.
Plattenlayout
Um alle Proben in einem Durchgang zu behandeln, wurden die Proben auf eine 96-Well-Platte verteilt. In einer 96-Well-Platte sind die 96 Wells (= Vertiefungen, um jeweils eine Probe aufzunehmen) in 12 Spalten (nummeriert von 1 bis 12) und 8 Zeilen (beschriftet mit Buchstaben von A bis H) angeordnet.
Die Wells am Rand der Platte blieben unbenutzt, d.h. es wurden 60 der 96 Wells benutzt.
Das Plattenlayout ist hier.
Plattenlayout: Spalten
Aus dem Plattenlayout erkennen wir, dass sich die 12 Spalten der 96-Well-Platte wie folgt unterscheiden:
Die Spalten 1 und 12 wurden nicht benutzt und blieben leer.
In den Spalten 2 bis 9 wurden jeweils T-Zellen und Target-Zellen zusammen gebracht (“Kokultur”), in den Spalten 10 und 11 wurden nur Targetzellen eingebracht (“Monokultur”). Letzteres diente dazu, zu sehen, ob die Targetzellen sich teilen, wenn sie nicht von T-Zellen behelligt werden.
Die Spalten 2 und 6 enthielten die T-Zellen des ersten Vergleichsprobanden (C1), die Spalten 3 und 7 die des Patienten (P1), die Spalten 4 und 8 die des zweiten Vergleichsprobanden (C2) und die Spalten 5 und 9 die des dritten Vergleichsprobanden (C2).
In allen Wells (außer den leeren Wells in Spalten 1 und 12) wurden jeweils 12000 Targetzellen ausgesäht. In den Spalten mit T-Zellen wurden entweder ebenso viele T-Zellen wie Target-Zellen ausgesäht (Spalten 2 bis 5) oder doppelt so viele (Spalten 6 bis 9).
In allen Spalten außer Spalte 11 wurde der Proliferationshemmer Aphidicolin zugegeben. Lediglich in Spalte 11 wurde er weg gelassen, so dass man durch Vergeich zwischen den Spalten 10 und 11 erkennen kann, ob Aphidicolin überhaupt wirkt.
Die folgende Tabelle fasst all dies zusammen:
Bedeutung der Tabellenspalten
plate_column: Index der Plattenspalteused: ob die Wells der Spalte benutzt oder leer gelassen wurdencontent: welche Zelltypen ins Well gesäht wurdensubject: Ursprung der T-Zellen: P1 für den Patienten, C1-C3 für die Vergleichsprobanden, NA für Wells ohne T-Zellenaphidicolin: ob die Targetzellen vorab mit dem Proliferations-Hemmer Aphidocolin behandelt wurdenn0_target-cells: Anzahl der Target-Zellen, die pro Well ausgesäht wurden (stets 12000, außer in leeren Wells)n0_target-cells: Anzahl der zytotoxischen T-Zellen, die pro Well ausgesäht wurden (12000, 24000 oder 0)
Plattenlayout: Zeilen
Die verschiedenen Zeilen unterscheiden sich dadurch, wie viel anti-CD3 auf die Targetzellen aufgebracht wurde:
# A tibble: 8 × 3
plate_row used conc_anti_cd3
<chr> <lgl> <dbl>
1 A FALSE NA
2 B TRUE 0
3 C TRUE 0
4 D TRUE 1
5 E TRUE 1
6 F TRUE 5
7 G TRUE 5
8 H FALSE NABedeutung der Tabellenspalten
plate_row: Index der Plattenzeileused: ob die Wells der Zeile benutzt oder leer gelassen wurdenconc_anti_cd3: Konzentration der Lösung aus Antikörpern gegen CD3 (in µg/ml), mit denen die Targetzellen behandelt wurden, um sie den T-Zellen “schmackhaft” zu machen.
Aufgabe: Erstellen Sie diese Tabelle
Daten
Die Platte wurde 18 Stunden inkubiert. Alle 2 Stunden wurde von jedem Well jeweils vier floureszenz-mikoskopische Aufnahmen gemacht, die vier verschiedene Bereiche im Well zeigen. In jedem mikroskopischen Bild wurde automatisch die Anzahl grün leuchtender Zellen (also, noch lebender Target-Zellen) gezählt.
Das automatische Mikroskop hat die folgende Datei mit den Zell-Zahlen erzeugt: GFP_object_count_perImage20221020withIL2.txt.
Aufgabe: Inspizieren die Datei mit einem Text-Viewer und/oder mit Excel.
Wir laden die Tabelle wie folgt:
read_delim( "data_on_git/GFP_object_count_perImage20221020withIL2.txt", delim="\t", skip=6, local=locale(decimal_mark=",") ) -> table_raw
table_raw# A tibble: 10 × 242
`Date Time` Elapsed `B2, Image 1` `B2, Image 2` `B2, Image 3` `B2, Image 4`
<chr> <dbl> <dbl> <dbl> <dbl> <dbl>
1 20.10.2022 1… 0 245 196 283 185
2 20.10.2022 2… 2 275 184 305 199
3 20.10.2022 2… 4 272 210 321 208
4 21.10.2022 1… 6 272 217 321 226
5 21.10.2022 3… 8 295 226 313 208
6 21.10.2022 5… 10 310 237 344 232
7 21.10.2022 7… 12 331 289 328 255
8 21.10.2022 9… 14 303 301 403 278
9 21.10.2022 1… 16 364 296 452 280
10 21.10.2022 1… 18 426 368 563 353
# ℹ 236 more variables: `C2, Image 1` <dbl>, `C2, Image 2` <dbl>,
# `C2, Image 3` <dbl>, `C2, Image 4` <dbl>, `D2, Image 1` <dbl>,
# `D2, Image 2` <dbl>, `D2, Image 3` <dbl>, `D2, Image 4` <dbl>,
# `E2, Image 1` <dbl>, `E2, Image 2` <dbl>, `E2, Image 3` <dbl>,
# `E2, Image 4` <dbl>, `F2, Image 1` <dbl>, `F2, Image 2` <dbl>,
# `F2, Image 3` <dbl>, `F2, Image 4` <dbl>, `G2, Image 1` <dbl>,
# `G2, Image 2` <dbl>, `G2, Image 3` <dbl>, `G2, Image 4` <dbl>, …Das skip=6 bewirkt, dass die ersten 6 Zeilen (die noch keine Daten enthalten) übersprungen werden sollen, und das locale-Argument löst das Problem, dass diese Datei mit einer finnischen Excel-Version gespeichert wurde, die (ebenso wie die deutsche) Kommas statt Punkte als Dezimaltrenner verwendet.
Aufgabe: Zur weiteren weiteren Bearbeitung hätten wir die Tabelle gerne in langer statt breiter Form. Formen Sie dazu die Tabelle zu folgender Form um:
# A tibble: 2,400 × 3
hours_incub name count
<dbl> <chr> <dbl>
1 0 B2, Image 1 245
2 0 B2, Image 2 196
3 0 B2, Image 3 283
4 0 B2, Image 4 185
5 0 C2, Image 1 278
6 0 C2, Image 2 224
7 0 C2, Image 3 174
8 0 C2, Image 4 189
9 0 D2, Image 1 168
10 0 D2, Image 2 207
# ℹ 2,390 more rowsDie Spaltennamen enthalten drei Bestandteile: Der Buchstabe gibt die Platten-Zeile an, die Zahl direkt danach die Platten-Spalte, und die Zahl am Schluss die Nummer des Bilders. Letzteres bezieht sich darauf, dass das Sichtfeld des Mikroskops deutlich kleiner ist als die Fläche eines Wells. Daher nimmt das automatische Mikroskop zu jedem Well jeweils 4 Bilder von 4 zufällig gewählten Ausschnitten auf und nummeriert diese von 1 bis 4.
Der folgende Code (den Sie nicht verstehen müssen) zerlegt die Spalte name in diese drei Bestandteile.
tbl_intermediate %>%
mutate( matches = as_tibble( str_match( name, "(.)(..?), Image (.)" ) ) ) %>%
unnest( matches ) %>%
select( hours_incub, plate_row = V2, plate_column = V3, image_no = V4, count ) %>%
mutate( image_no = as.integer(image_no) ) %>%
mutate( plate_column = as.integer(plate_column) ) -> tbl_long
tbl_long# A tibble: 2,400 × 5
hours_incub plate_row plate_column image_no count
<dbl> <chr> <int> <int> <dbl>
1 0 B 2 1 245
2 0 B 2 2 196
3 0 B 2 3 283
4 0 B 2 4 185
5 0 C 2 1 278
6 0 C 2 2 224
7 0 C 2 3 174
8 0 C 2 4 189
9 0 D 2 1 168
10 0 D 2 2 207
# ℹ 2,390 more rowsDer Einfachkeit halber addieren wir die Zellzahlen der jeweils vier Bilder einfach auf, so dass wir eine Zellzahl pro Well und Zeitpunkt haben. (Eigentlich sollten wir vorher überprüfen, ob die vier Zahlen jeweils nicht zu weit voneinader abweichen; das überspringen wir aber diesmal.)
Aufgabe: Addieren Sie die Zellzahlen der jeweils vier Bilder pro Well und Zeitpunkt. Ihr Ergebnis sollte so aussehen:
# A tibble: 600 × 4
# Groups: hours_incub, plate_row [60]
hours_incub plate_row plate_column count
<dbl> <chr> <int> <dbl>
1 0 B 2 909
2 0 B 3 981
3 0 B 4 794
4 0 B 5 964
5 0 B 6 907
6 0 B 7 914
7 0 B 8 781
8 0 B 9 1007
9 0 B 10 817
10 0 B 11 944
# ℹ 590 more rowsMonokulturen
Nun betrachten wir zunächst nur die Spalten 10 und 11, die Targetzellen, aber kein T-Zellen enthalten. Hier ist ein Plot dieser Daten:
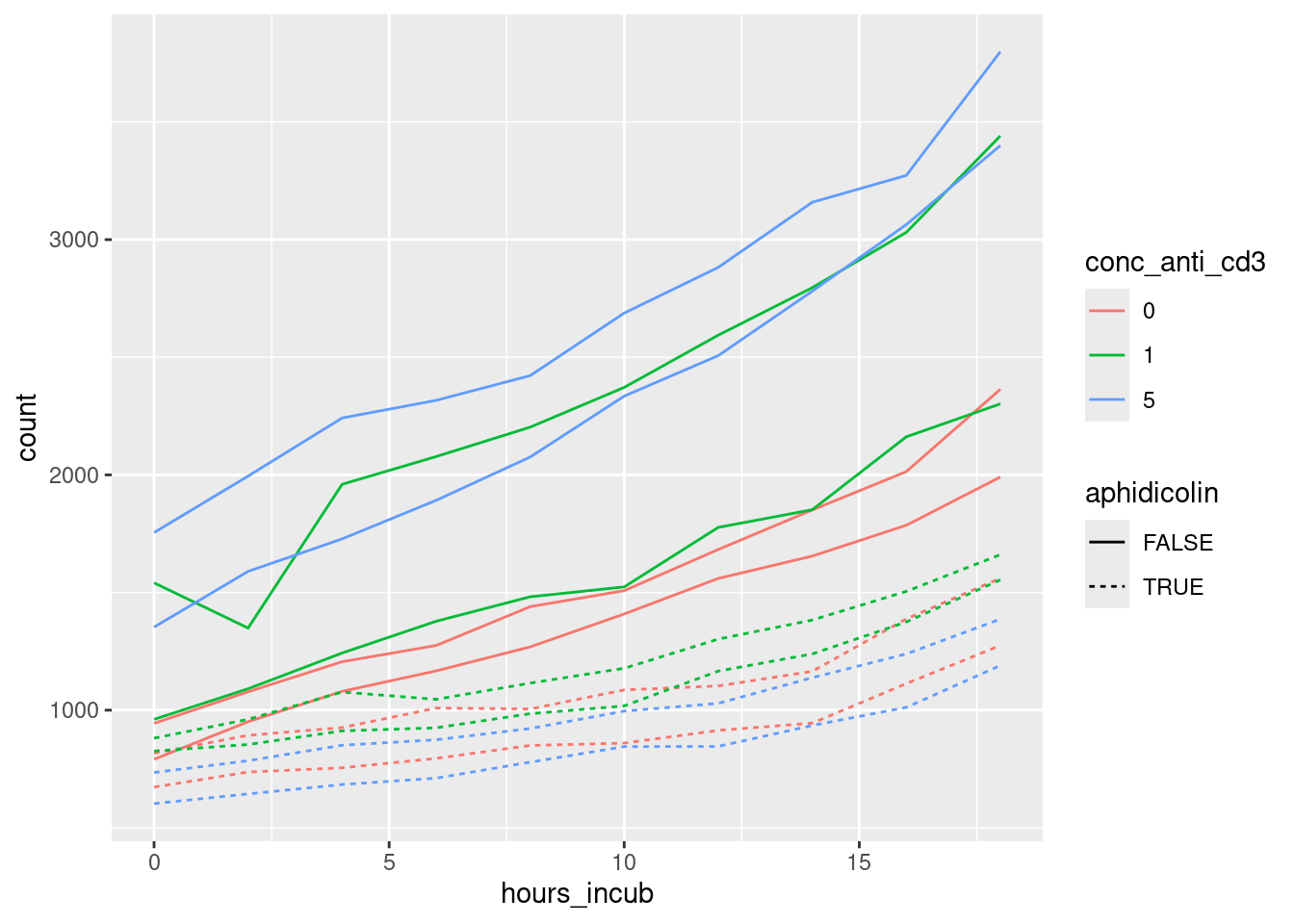
Aufgabe: Erstellen Sie diesen Plot.
Hinweise dazu: Filtern Sie die Tabelle zunächst so, dass Sie nur die Plattenspalten mit Monokultur (Spalte content der Tabelle plate_cols ist "target cells only") haben, sowie natürlich nur die Zeilen, die nicht am Rand liegen und somit benutzt wurden (Spalte used der Tabelle plate_rows ist TRUE).
Plotten Sie dann eine Linie für jedes Well. (Hinweis: Sie müssen die group-aesthetic verwenden, um anzugeben, welche Tabellenzeile zur selben Linie des Plots gehören. Dazu können Sie sich mit mutate( well=str_c( plate_row, plate_column) ) eine Spalte erzeugen, die für jedes Well einen anderen Wert hat.)
Uns fällt auf dass die Wells zum Zeitpunkt 0 nicht alle dieselbe Zellzahl haben (obwohl ja eigentlich jedes Well anfangs dieselbe Zahl an Zellen (nämlich 12000) haben sollte). Um die Änderung mit der Zeit gut beurteilen zu können, wäre es hilreich, alle Zahlen durch den Wert bei Zeitpunkt 0 zu teilen.
Aufgabe: Erstellen Sie die folgende Tabelle, die nur die Zellzahlen zum Zeitpunkt 0 enthält:
tbl_counts_at_zero# A tibble: 60 × 3
plate_row plate_column count_at_0
<chr> <int> <dbl>
1 B 2 909
2 B 3 981
3 B 4 794
4 B 5 964
5 B 6 907
6 B 7 914
7 B 8 781
8 B 9 1007
9 B 10 817
10 B 11 944
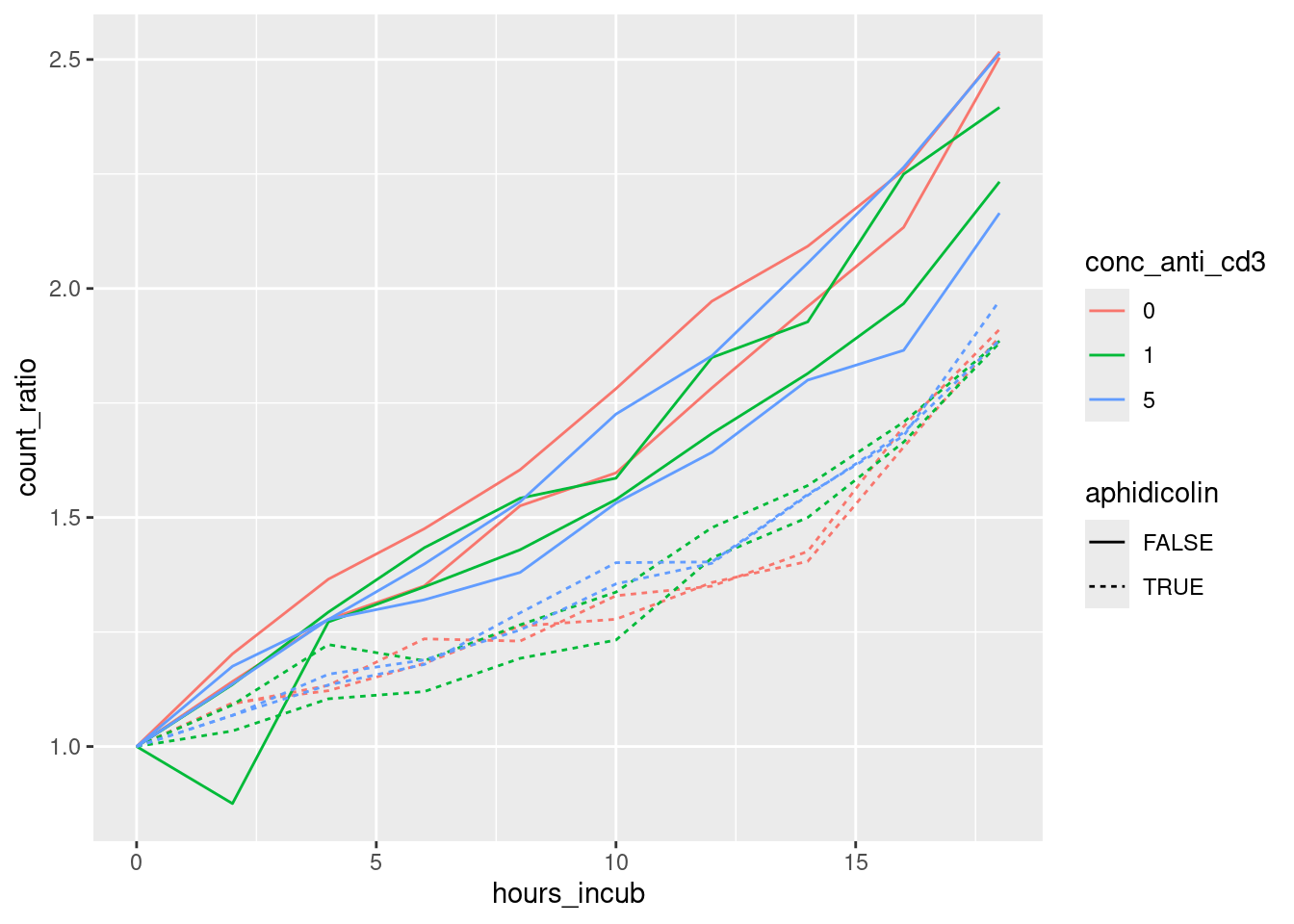
# ℹ 50 more rowsAufgabe: Teilen Sie in der langen Tabelle jede Zahl durch die Zellzahl zum Zeitpunkt 0.
Aufgabe: Erstellen Sie nun denselben Plot wie zuvor, verwenden aber als y-Achse nun die Verhältnisse der Zellzahlen zum Zeitpunkt 0:
Aufgabe: Skalieren Sie nun noch die y-Achse logarithmisch
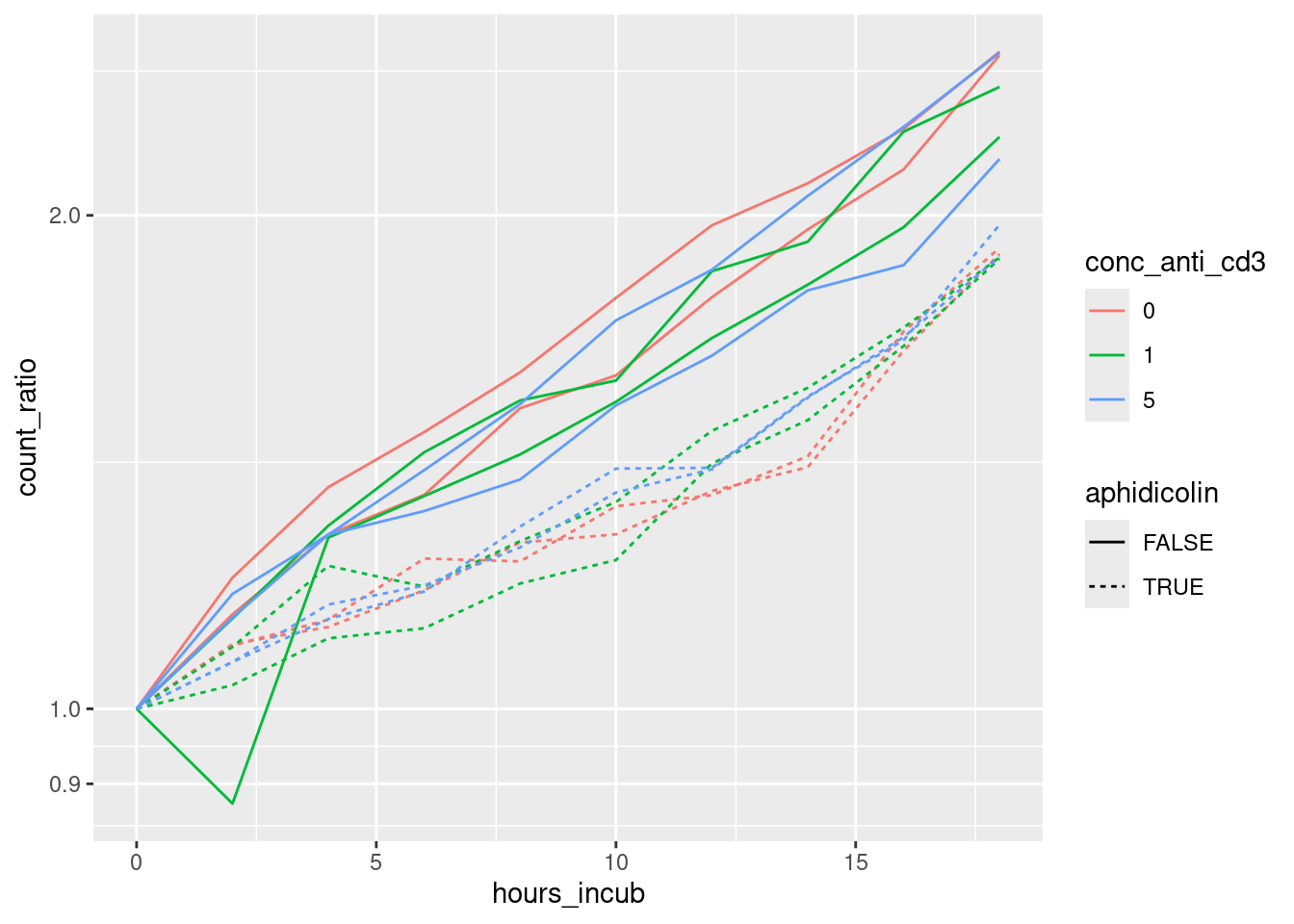
Aufgabe: Interpretieren Sie den Plot. Was erkennen Sie?
Mögliche Fragen: Hat die Belegung mit anti-CD3 einen Einfluss auf die Vermehrung der Zellen? Hat das Aphidicolin eine Wirkung? Wenn ja, wie stark ist sie?
Kokulturen
Der folgende Plot zeigt nun die Daten der Kokulturen. Die Facetten-Spalten sind die vier Probanden, die Facetten-Zeilen die beiden Verhältnisse an T-Zellen zu Targetzellen.
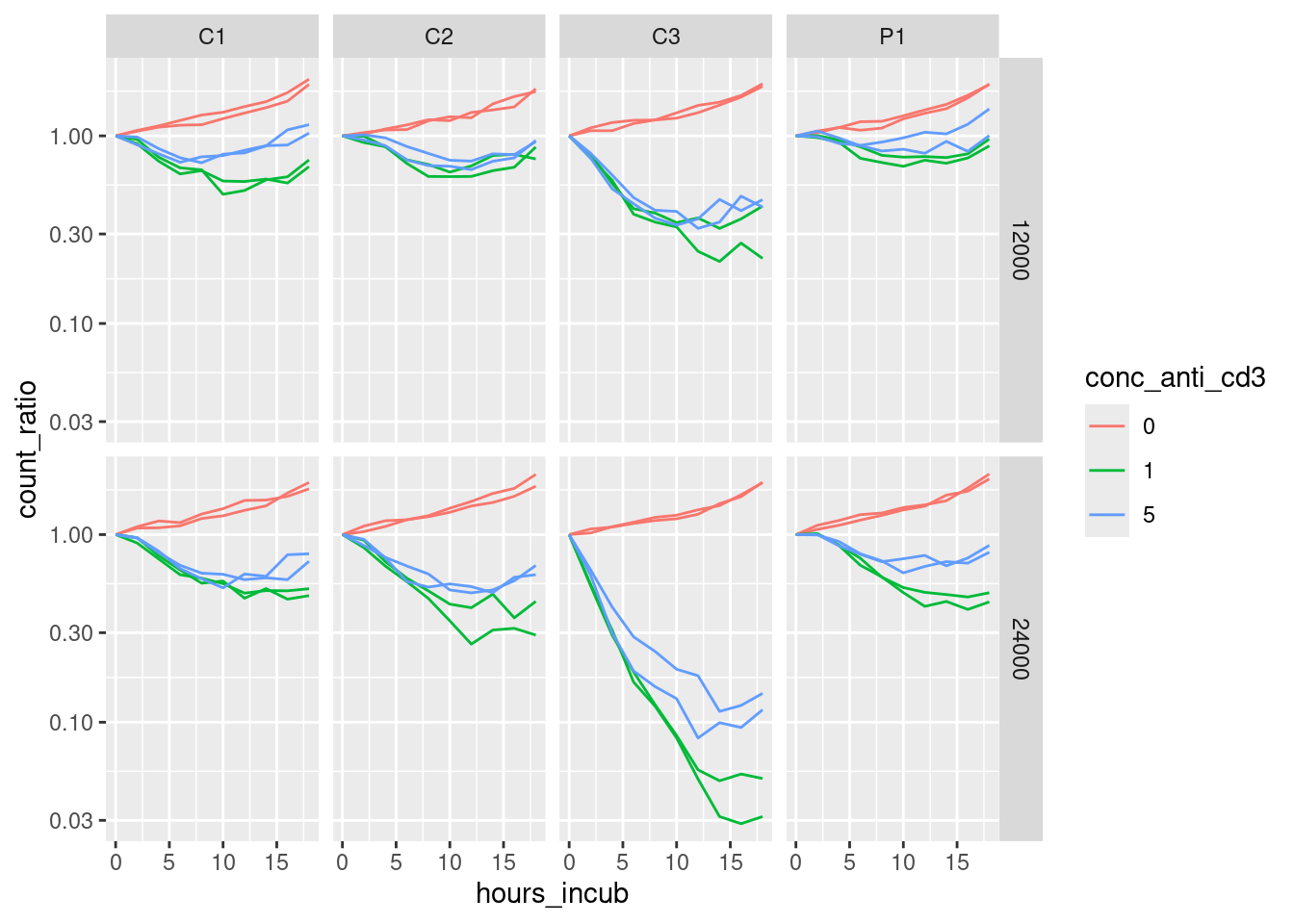
Aufgabe: Erstellen Sie diesen Plot.
Aufgabe Interpretieren Sie den Plot.