Vorlesung “Datenanalyse in der Biologie”: Einführung
Wintersemester 2023/23
Simon Anders, BioQuant, Universität Heidelberg
Einführendes Beispiel
Um Ihnen ein Gefühl dafür zu geben, was Sie hier in diesem Semester lernen werden, zeige ich Ihnen im Folgenden ein Beispiel einer typischen Datenanalyse.
Am Ende des Semesters werden Sie solche Analysen selbst duchführen können.
Problemstellung
Zu Beginn der Covid-Pandemie untersuchten wir an ZMBH, ob sich das sog. LAMP-Verfahren als Alternative zur PCR eignet, um das SARS-Cov2-Virus nachzuweisen.
LAMP ist, wie PCR, ein Verfahren, das DNA mit Polymerase vervielfacht, wenn passende Primer vorliegen. Es ist nicht so präzise, aber technisch einfacher und billiger.
Bei der DNA-Amplifikation werden Protonen freigesetzt, daher kann man das Ergebnis durch Messen des pH-Werts auslesen. Wir haben dazu Phenolrot zugegeben, um
Wir hatten 96 klinische Proben (Rachenabstriche), die bereits vom Labor der Uniklinik mit PCR getestet wurden. Hier ist ein Bild der Platte:
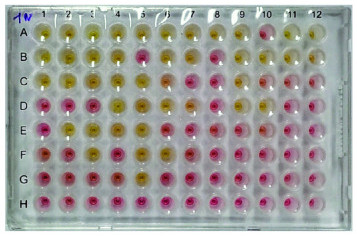
Wells, in denen DNA durch LAMP vervielfältigt wurde, sind von rot auf gelb umgeschlagen.
Ergebnis
Inkubationskurven
Um die Farbe der Wells zu messen, haben wir einen Platten-Scanner verwendet, um die Absorption von Licht bei zwei Wellenlängen (434 nm [blau] and 560 nm [gelb]) zu messen. Da wir nicht wussten, wie lange man inkubieren muss, haben wir die Platte alle 5 Minuten auf Eis gelegt und mit dem Scanner gemessen, dann wieder erwärmt. Hier sind die 96 Kurven:
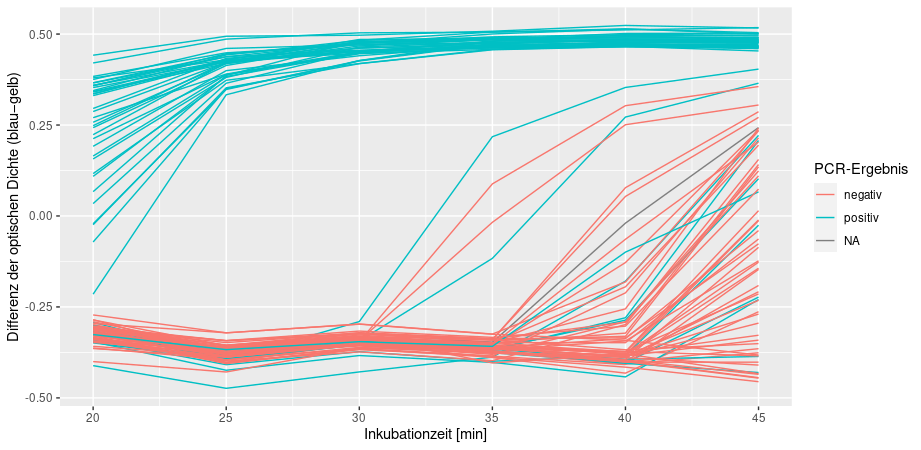
- Jeder Kurve (Linie) steht für eine Probe, d.h., ein Well der 96-Well-Platte
- Die Farben der Kurven zeigend as Ergebnis des PCR-Tests des Klinik-Labors
- Die x-Achse ist die Inkubationszeit. Alle 5 Minuten wurde gemessen (Knicks in den Kurven)
- Die y-Achse ist das Messergebnis (Ausgabe des Platten-Scanners). Negative Werte bedeuten rote, psotive bedeuten gelbe Farbe.
Wir erkennen:
- Die Proben, die laut PCR SARS-CoV-2-positiv waren, schlagen auf rot um. Ab 30 Minuten sind alle Kurven umgeschlagen.
- Ab 35 oder 40 min beginnen auch negative Proben umzuschlagen.
-> Wir verwenden also das Messergebnis bei 30 Minuten
Vergleich LAMP vs PCR
Der nächste Plot zeigt einen Vergleich der Ergebnisse der beiden Analyse-Verfahren:
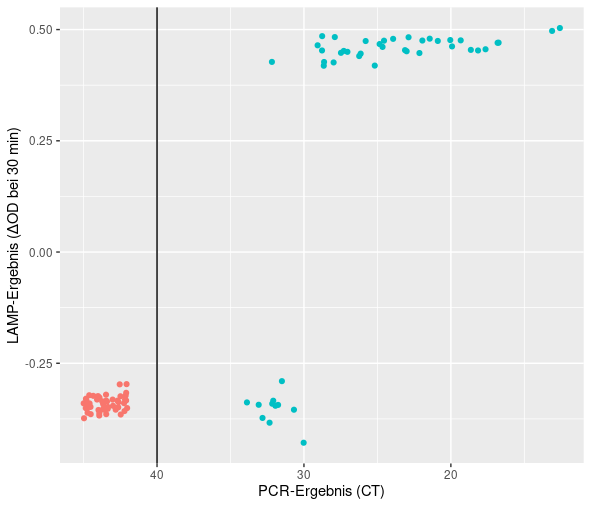
In diesem Streudiagramm (scatter plot) gilt: - Jeder Punkt ist eine Probe - Die x-Achse zeigt das Ergebnis der qPCR. Angegeben ist der “threshold cycle” (\(c_T\)), d.h., die PCR-Runde, in der das Signal erstmals den gesetzten Detektions-Schwellwert überschritt. Wenn nach 40 Runden noch nichts zu sehen war, wurde die Probe als negativ gewertet (rote Punkte links der schwarzen Linie). - Die y-Achse ist wieder das LAMP-Ergebnis; diesmal nur der Wert bei 30 min.
Sensitivität und Spezifität
Nun können wir ablesen, wie gut der LAMP-Test funktioniert hat:
| PCR_res | LAMP_res | n |
|---|---|---|
| negative | negative | 48 |
| positive | negative | 11 |
| positive | positive | 36 |
| NA | negative | 1 |
Von insgesamt 47 PCR-positiven Proben hat LAMP 36 korrekt als positiv erkannt. Die Sensitivität betrugt also 36/47=77%.
Von insgesamt 48 negativen Proben wurden 48 korrekt als negativ erkannt. Die Spezifität betrug also 100%.
Roh-Daten
Um diese Plots zu erstellen, hatten wir folgende Daten:
Platten-Scans
Der Platten-Scanner hat Tabellen als Excel-Dateien ausgegeben. Hier ist ein Beispiel: Excel-Datei vom Tecan-Scanner
Wir haben ein Arbeitsblatt (worksheet) pro Messung. Darin finden sich zwei Blöcke mit Messwerten (für die beiden Wellenlängen), in denen die Werte so angeordnet sind, wie die Wells in der Tabelle liegen.
PCR-Ergebnisse
Außerdem gab es eine Tabelle mit PCR-Ergebnissen, die so aussah:
| plate | well | CT |
|---|---|---|
| CP00001 | A1 | 12.58 |
| CP00001 | A2 | 27.29 |
| CP00001 | A3 | 18.64 |
| CP00001 | A4 | 16.82 |
| CP00001 | A5 | 24.65 |
| CP00001 | A6 | 18.15 |
| CP00001 | A7 | 25.18 |
| CP00001 | A8 | 26.24 |
| … | … | … |
Was war zu tun?
Um die Plots zu erstellen, waren diese Schritte erforderlich:
- Die Tabellen vom Scanner vom 2D-Platten-Format (8 Zeilen x 12 Spalten) in ein langes Format (96 Zeilen) umformen
- Die beiden Blöcke nebeneinander setzen
- Zeitpunkte aneinander fügen
- In jeder Zeile das zugehörige PCR-Ergebnis hinzufügen
Dann hat man eine “Master-Tabelle” mit 6x96 Zeilen wie folgt:
| time | row | column | CT | absBlue | absYellow |
|---|---|---|---|---|---|
| 20 | A | 1 | 12.58 | 1.5294 | 1.1088 |
| 20 | A | 2 | 27.29 | 1.5387 | 1.3152 |
| 20 | A | 3 | 18.64 | 1.4276 | 1.0480 |
| 20 | A | 4 | 16.82 | 1.3988 | 1.0152 |
| 20 | A | 5 | 24.65 | 1.3980 | 1.1025 |
| 20 | A | 6 | 18.15 | 1.4120 | 1.0699 |
| 20 | A | 7 | 25.18 | 1.3123 | 1.1957 |
| 20 | A | 8 | 26.24 | 1.3827 | 1.1912 |
| 20 | A | 9 | 30.02 | 1.0943 | 1.5057 |
| … | … | … | … | … | … |
| 25 | E | 1 | Inf | 1.2868 | 1.6817 |
| 25 | E | 2 | 16.75 | 1.4493 | 0.9888 |
| 25 | E | 3 | 20.04 | 1.4393 | 1.0016 |
| 25 | E | 4 | 30.68 | 1.2604 | 1.6517 |
| 25 | E | 5 | 23.93 | 1.4447 | 1.0151 |
| 25 | E | 6 | Inf | 1.2787 | 1.6454 |
| 25 | E | 7 | Inf | 1.1582 | 1.5146 |
| 25 | E | 8 | Inf | 1.1465 | 1.5343 |
| … | … | … | … | … | … |
| 45 | H | 9 | Inf | 1.3437 | 1.2698 |
| 45 | H | 10 | Inf | 1.3691 | 1.1659 |
| 45 | H | 11 | Inf | 1.2609 | 1.6431 |
| 45 | H | 12 | Inf | 1.3004 | 1.5087 |
Damit können wir die Plots zeichnen, indem wir dem Computer mitteilen, welche Spalten für welche Plot-“Koordinaten” (x, y, Farbe) benuzt werden sollen.
Wie macht man das?
Mögliche Werzeuge:
Tabellenkalkulations-Programme (spreadsheet applications) wie Microsoft Excel
Programme, um wissenschaftliche Plots zu erstellen, z.B. Origin
statistische Programmiersprachen, wie z.B. R.
Wir werden R erlernen.
Bioinformatik: Was ist das?
Typische Aufgaben in Bioinformatik, data science und Statistik:
Aufbereitung von Daten (data preprocessing): Bevor man mit Daten arbeiten kann, muss man sie in eine geeignete Form bringen, meist in die Form einer Tabelle.
Datenreduktion (data reduction), beschreibende Statistik (descriptive statistics): Wir versuchen, große Datenmengen geeignet zusammen zu fassen, z.B. in dem wir Mittelwerte oder andere summary statistics bestimmen.
Datenvisualisierung (data visualization) / “Plotting”: Darstellung (tabellarischer) Daten in graphischer Form (als Plot).
explorative Datenanalyse (exploratory data analysis, EDA): Wir haben Mess- oder andere Daten und suchen darin nach Mustern, Gemeinsamkeiten, oder anderem.
Schließende Statistik (inferential statistic): Wir verwenden statistische Hypothesentests (statistical tests), um zu ermitteln, ob ein Ergebnis wirklich “echt” ist oder “nur durch Zufall” zustande gekommen ist.
Außerdem gehört zur Bioinformatik der Umgang mit DNA-Sequenzen:
Datenbanken von Genom-Sequenzen
Suche von (exakt gleichen oder zumindest verwandten) Sequenzen
Vergleich von Sequenzen, um evolutionäre Verwandtschaft zu bestimmen (Phylogenie, molecular clock)
sequenzier-basierte Assays, z.B. RNA-Seq und verschiedene Assays zur Epigenetik
usw.
Außer Sequenzierdaten und Meßwerten gibt es auch andere Datentypen, z.B. Mikrokopie-Bilder
Zum Grundwerkzeug gehört, mit Datentabellen zu arbeiten. Daher lernen wir R.
Was ist R?
R ist eine “statistische Programmier-Umgebung”.
Andere solche Werzeuge sind z.B. SAS und SPSS.
In der Biologie wird meist R verwendet, in der Medizin gerne SAS, in anderen Fächern SPSS.
R is kostenlos und “open source”.
Es gibt eine Vielzahl von Erweiterungspaketen (“packages”), die spezielle Funktionalitäten bereit stellen
Zum bequemen Arbeiten gibt es eine “Arbeits-Umgebung”, genannt “RStudio”
Installation von R
Um die Übungen in diesem Kurs machen zu können, installieren Sie bitte auf Ihrem Laptop:
Sie brauchen keinen leistungsfähigen Rechner; auch ein 10 jahre alter Laptop genügt meist.
Literatur
Diese Lehrbücher, die alle sowohl gedruckt, wie auch kostenlos online verfügbar sind, können Sie neben der Vorlesung nutzen, um etwas nachzuschlagen:
Erste Schritte mit R
siehe nächstes Dokument
Nur Übung macht den Meister
Datenanalyse mit R ist ein Handwerk. Man lernt es durch Üben, nicht, indem man in der Vorlesung nur zuhört. Wenn Sie beim Üben zurück fallen, werden Sie schnell nicht mehr folgen können. Machen Sie also die Hausaufgaben!
Programmieren kann fristrieren: Oft hängt man an einem kleinen Fehler, den man einfach nicht sieht, oder hat etwas leicht falsch verstanden, und dann versteht einen der Computer deshlab nicht. Mit einem Tipp kommt man aber sofort weiter. Daher brauchen Sie, besonders anfangs, beim Üben einen Tutor!
Daher werden wir während der Vorlesung immer wieder Pausen machen, während derer Sie im Hörsaal üben und sich an die anwesenden Tutoren wenden können. Bringen Sie daher bitte Ihren Laptop stets mit. (Wenn Sie keinen Laptop besitzen, wenden Sie sich bitte an mich.) Laden Sie bitte vorher den Akku auf.
Damit Sie von den Tutoren profitieren, müssen Sie sich trauen, Fragen zu stellen. Das ist extrem wichtig.
Hausaufgabe
Bitte machen Sie bis Donnerstag bitte folgendes:
Installieren Sie auf Ihrem Laptop R und RStudio (s.o.).
Stellen Sie R einige einfache Rechenaufgaben, z.B. \(3+7\), \(\frac{2\cdot 5-13}{3}\), \(\sqrt{3}\), usw.
Was ist \(\sqrt{2}\sin\frac{pi}{4}\)? (Um \(\pi\) einzugeben, schreiben Sie einfach
pi.)Zeichnen Sie mit TurtleGraphics ein rechtwinkliges und ein gleichseitiges Dreieck.
Wählen Sie in RStudio mit dem Menü “Datei”, Unterpunkt “Neue Datei”, dann “Neues R-Skript”. Sie erhalten
ein leeres “Blatt”. Tippen Sie in dieses die Befehle für das Dreieck. Führen Sie den Code aus. Speichern Sie ihn dann ab, schließen Sie RStudio, starten es neu, und öffnen Sie Ihre Datei. Funktioniert der Code noch? Wenn nein, was fehlt?
Wenn Sie Schwierigkeiten haben, kein Sorge! WIr besprechen alles am Donnerstag nochmal Schritt für Schritt.
Webseite zur Vorlesung:

https://papagei.bioquant.uni-heidelberg.de/simon/Vl2324/