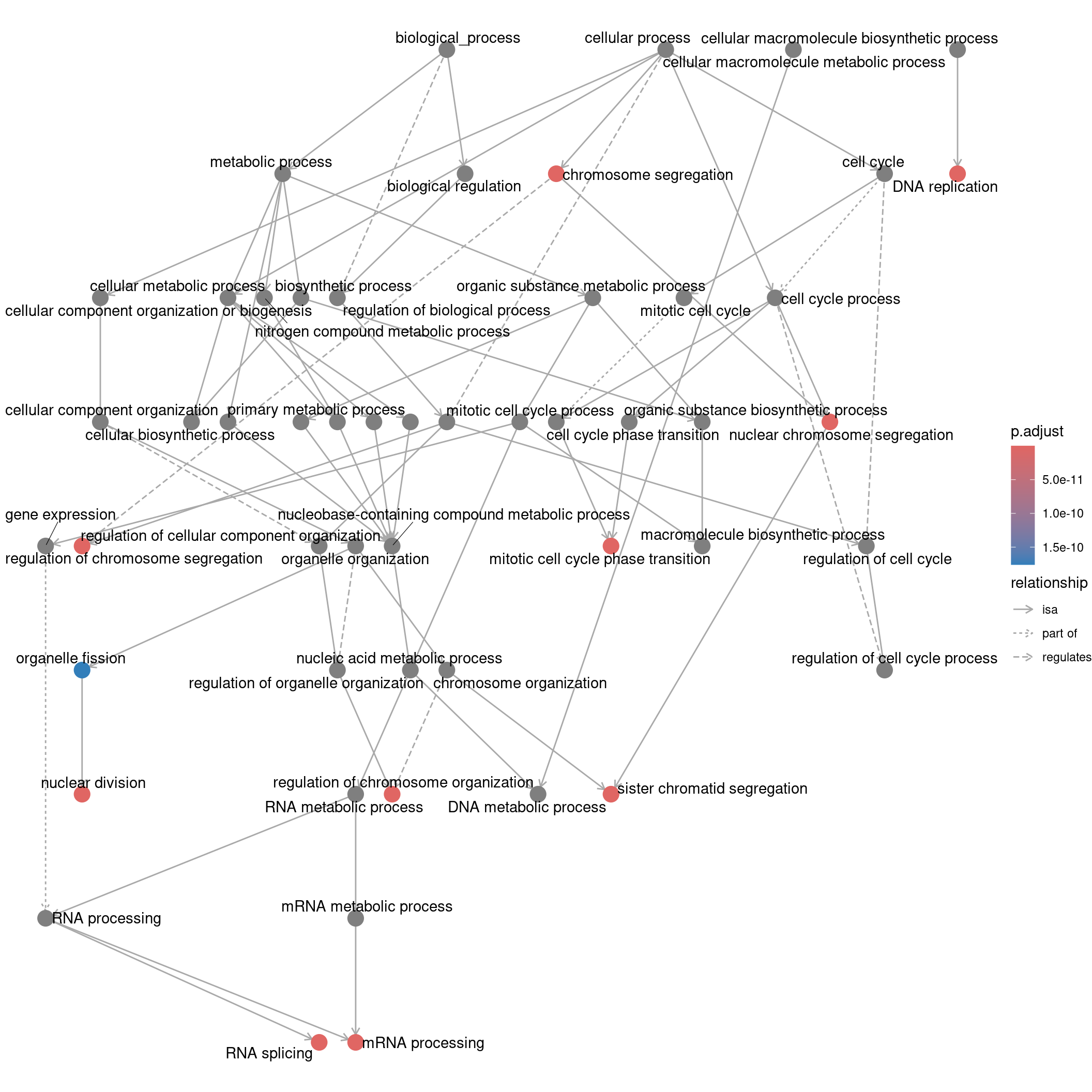
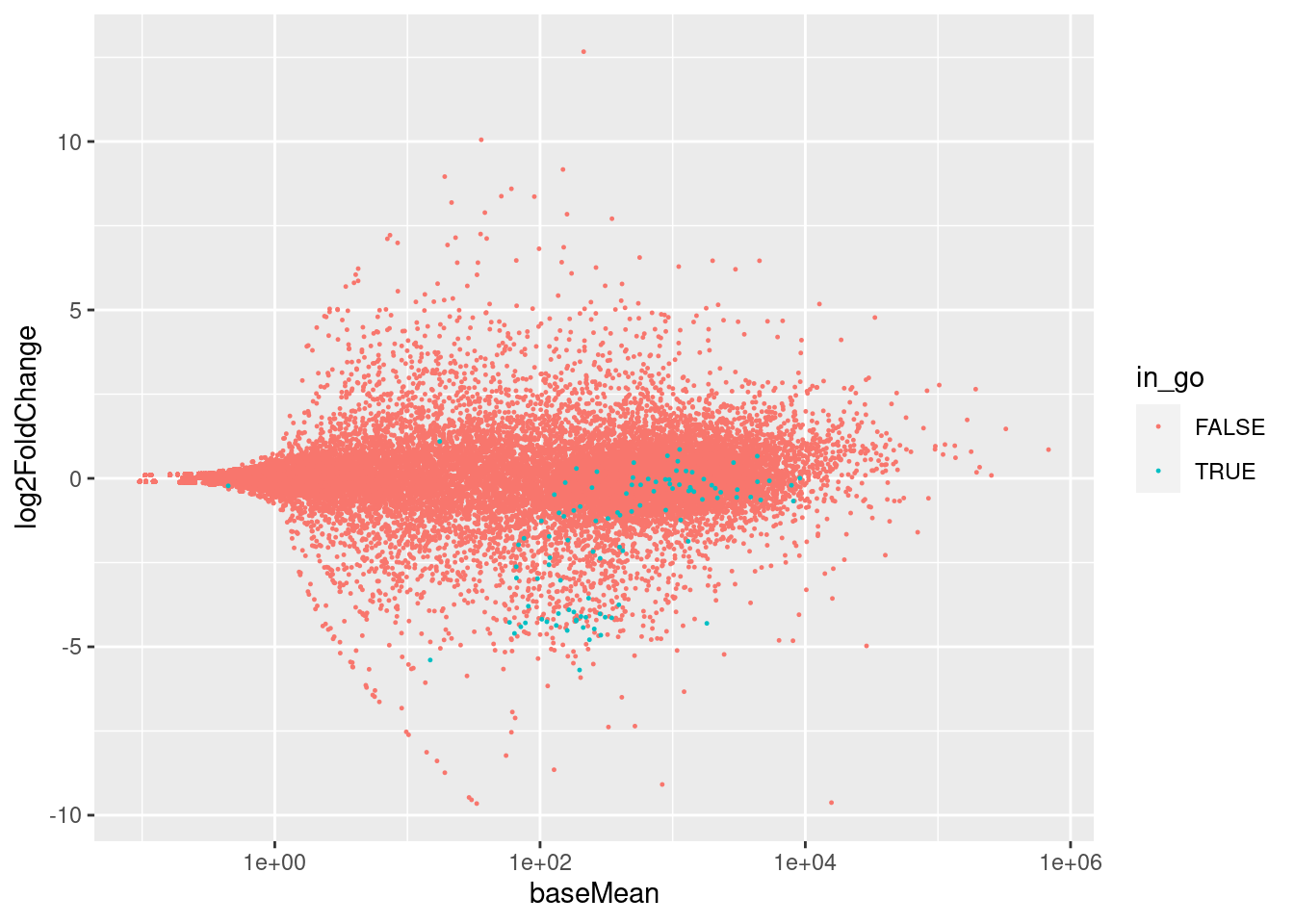
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value
Warning in nbinomGLM(x = x, Y = YNZ, size = size, weights = weightsNZ, offset =
offsetNZ, : the line search routine failed, unable to sufficiently decrease the
function value